H3K27me3是核心组蛋白H3的第27个氨基酸赖氨酸的氨基(N)末端尾部发生的组蛋白甲基化��,是最为常见的组蛋白甲基化修饰之一����。H3K27me3是具有组蛋白甲酰基转移酶活性的多梳抑制复合体2 (Polycomb Repressive Complex 2,PRC2)的下游靶标���,PRC2在干细胞多能性����、体细胞分化和增殖中具有不同的作用����,PRC2的酶亚基EZH1和EZH2催化H3K27的甲基化��,H3K27的甲基化是抑制基因转录的关键介质���,参与多个重要的生物学过程����。H3K27me3作为一种基因转录抑制因子�����,在胚胎发生和肿瘤形成中具有多种作用����。H3K27me3的缺失发生在恶性周围神经鞘膜瘤(MPNST)的一个重要亚群中���,其他肿瘤也可能表现为H3K27me3表达缺失���,如胚胎性横纹肌肉瘤���、脑膜瘤���、放射相关性未分类肉瘤���、放射相关性血管肉瘤��、去分化软骨肉瘤����、黑色素瘤和默克尔细胞癌等�����。因此H3K27me3可以作为恶性周围神经鞘膜瘤的鉴别指标���。
H3K27me3抗体是EpiCypher最新研究的SNAP-Certified™抗体之一���,并通过了独有的SNAP-Certification技术进行验证���,符合CUT&RUN和CUT&Tag应用中特异性和有效靶标富集的标准���。
产品详情
产品货号 | 13-0055 |
产品名称 | H3K27me3 Antibody, SNAP-Certified™ for CUT&RUN and CUT&Tag |
规格 | 100 µg |
产品类型 | Monoclonal [2084-1G5] |
宿主 | Rabbit |
浓度 | 0.5 mg/mL |
反应种属 | Human, Wide Range(Predicted) |
实验应用 | CUT&RUN, CUT&Tag |
免疫原 | A synthetic peptide corresponding to histone H3 trimethylated at lysine 27 |
保存温度 | Stable for 1 year at 4°C from date of receipt |
配方 | Antigen affinity-purified antibody in Borate buffered saline pH 8.0, 0.09% sodium azide |
建议稀释度 | 0.5 µg per reaction for CUT&RUN and CUT&Tag |
实例分析
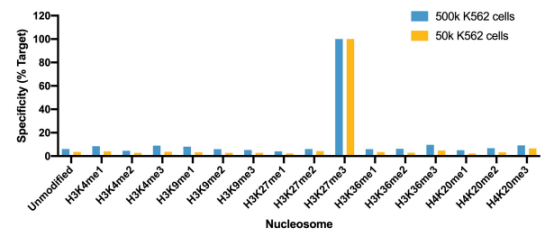
图1. CUT&RUN中SNAP特异性分析
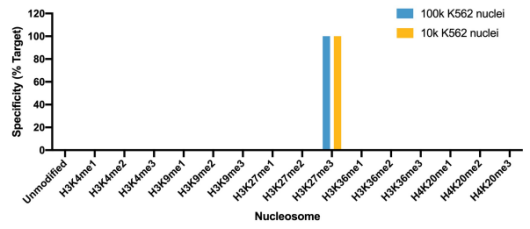
图2. CUT&Tag中SNAP特异性分析
使用SNAP-CUTANA™ K-MetStat Panel对H3K27me3抗体进行分析���,其与相关组蛋白PTMs的交叉反应性<20%���。
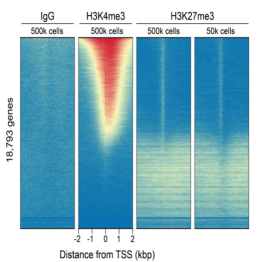
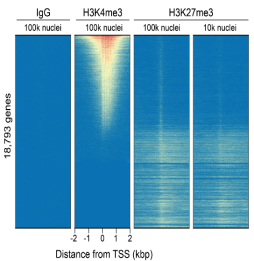
图3. CUT&RUN和CUT&Tag全基因组富集
H3K27me3与基因的抑制有关��,而H3K4me3一般在转录起始位点附近富集����,来激活基因转录����。
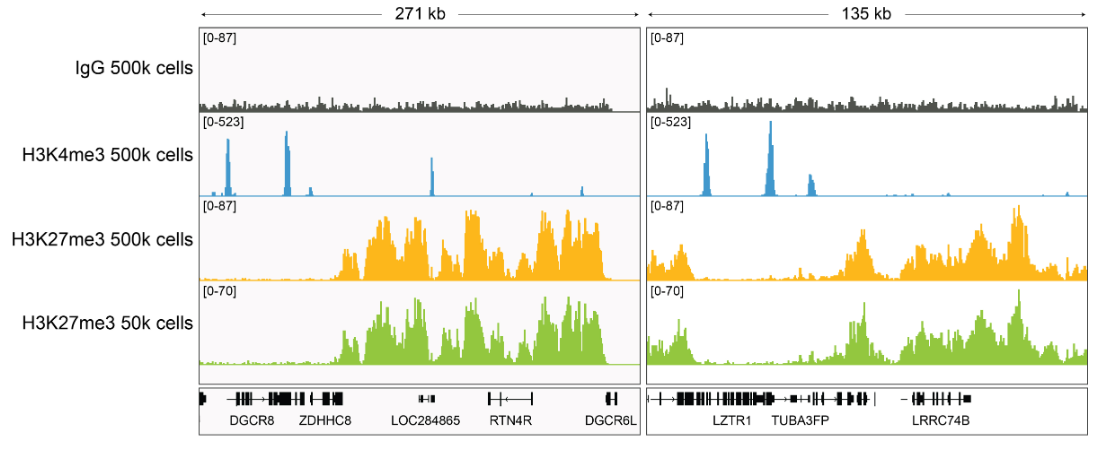
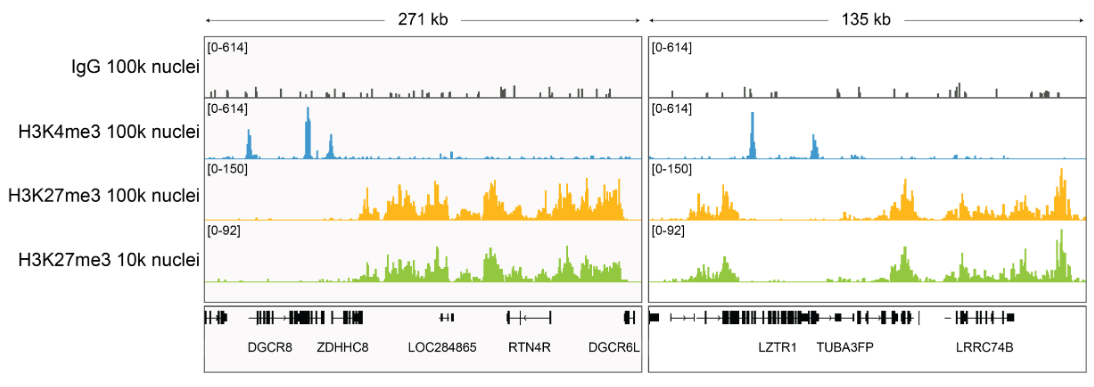
图4. H3K27me3抗体CUT&RUN和CUT&Tag峰图
高效抗体在代表性基因上表现出相似的峰值结构���,即使在细胞数量减少的情况下���,全基因组信号也高度稳定���。
H3K27me3抗体在CUT&RUN和CUT&Tag应用中符合EpiCypher特定批次SNAP-Certified™标准�����,不同的细胞数量下�����,一致的基因组富集证实了其高靶向效率���。SNAP-Certified™抗体具有交叉反应性低���、CUT&RUN和CUT&Tag性能稳定����、改善低细胞数的PTM分析等特点��。
关于EpiCypher
EpiCypher是一家成立于2012年的表观遗传学公司��。从专有组蛋白肽阵列平台EpiGold™开始����,EpiCypher开发了一系列同类产品����。同时���,EpiCypher是重组核小体制造和开发的全球领导者����。利用其独有技术��,不断添加高纯度修饰重组核小体(dNucs™)产品��。dNuc™多样性的产品为破译组蛋白编码和加速药物开发提供了强大的工具����。
EpiCypher还将dNuc™技术广泛的应用于多种分析测定产品中����,包括���:SNAP-ChIP®Spike-in Controls(用于抗体分析和ChIP定量)����, EpiDyne®底物(用于染色质重塑和抑制剂筛选及开发)���,dCyher™测定(用于探究表观遗传蛋白质-组蛋白PTM结合相互作用)��。最近����,EpiCypher还推出了针对ChIC���、CUT&RUN和CUT&Tag的高灵敏度表观基因组图谱CUTANA™分析����。

如需了解更多详细信息或相关产品���,请联系EpiCypher中国授权代理商-欣博盛生物
全国服务热线: 4006-800-892 邮箱: market@qckc0531.com
深圳: 0755-26755892 北京: 010-88594029
广州����:020-87615159 上海: 021-34613729
代理品牌网站: www.qckc0531.com
自主品牌网站: www.neobiosescience.net